高通量测序(NGS)平台具有高输出量且准确的测序性能,已大规模用于识别恶性肿瘤相关的异常DNA甲基化并在辅助诊断、预后预测和病情监测等临床应用方面发挥重要作用。随着过去二十年的快速发展,基于桥式扩增和合成测序(SBS)原理的因美纳NGS测序仪(HiSeq2500, NovaSeq6000, HiSeqX10等)已成为使用最广泛的NGS平台。近年来,华大智造(MGI)推出了基于DNA纳米球(DNBs)扩增和结合引物锚定合成(cPAS)技术的新型测序仪(MGISEQ-2000、DNBSEQ-G99、DNBSEQ-T7等)也表现出优秀的输出性能,并逐渐成为NGS测序的新选择。近日,8455新葡萄娱集团am对比了华大智造MGISEQ-2000测序平台和因美纳NovaSeq 6000测序平台用于亚硫酸氢盐靶向测序的性能。该研究已发表在Clinical Epigenetics(IF 5.7)上[1]:“Cross-platform comparisons for targeted bisulfite sequencing of MGISEQ-2000 and NovaSeq6000(亚硫酸氢盐靶向测序的跨平台对比:MGISEQ-2000 VS NovaSeq 6000)”

DNA甲基化已成为临床应用的重要分子生物标记物。亚硫酸氢盐测序是目前检测甲基化的通用标准技术,可以在单个碱基对分辨率下定量检测DNA甲基化。目前亚硫酸氢盐测序主要是在因美纳测序平台上进行开发和验证的,而尚未在华大智造平台上进行全面测试。在本研究中,我们使用8455新葡萄娱集团am自主研发的无创胰腺癌早检产品PDACatch同时在华大智造MGISEQ-2000和因美纳NovaSeq6000进行cfDNA亚硫酸氢盐靶向测序,并在测序质量、甲基化一致性、检测灵敏性和临床准确性等方面,系统地比较了两个测序平台之间的性能差异。
研究结果
1、华大智造MGISEQ-2000平台进行亚硫酸氢盐靶向测序的性能良好
我们制备了一个人类全基因组序列(WGS)文库作为亚硫酸氢盐测序的对照。为了生成亚硫酸氢盐(BS)目标文库,我们将完全甲基化的基因组NDA(meDNA)以0、0.002、0.01、0.02和0.05的比例稀释到B淋巴母细胞来源的基因组DNA(NA12878)中,并使用MethylTitan方案制备文库。然后在MGISEQ-2000上,使用四种不同的平衡文库掺入比例(50%,30%,10%和0% WGS文库)对BS文库进行测序。

图1. MGISEQ-2000采用不同对照文库进行亚硫酸氢盐靶向测序并检测其测序质量的实验设计
在测序质量方面,我们发现在未掺入WGS文库生成的数据中,高质量reads (Phred评分≤30)的百分比明显低于其他数据(图2)。与掺入不同WGS文库比例生成的数据相比,使用30% WGS库生成的测序数据显示出略低的测序错误率(图3)。

图2. 甲基化文库的高质量测序比例
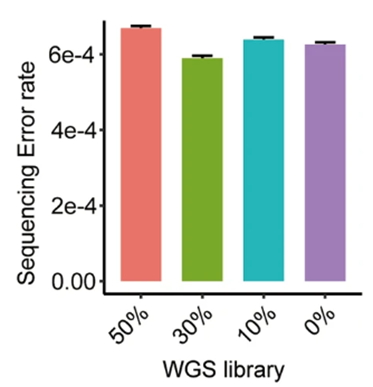
图3. 不同WGS文库比例生成的BS测序错误率
此外,我们评估了在MGISEQ-2000上对不同对照文库进行亚硫酸氢盐测序的定量准确性。结果表明,在不同对照文库生成的4个数据集中,估计的meDNA比率与期望的比率具有良好的一致性(R2 = 0.95)。我们还发现估计的meDNA比率略高于预期,这表明MGISEQ-2000可能检测到比预期更高的甲基化水平(图4)。
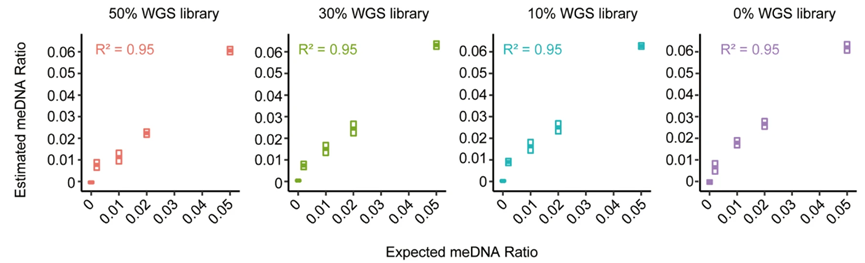
图4. 估计的完全甲基化基因组比率与期望值高度一致
2、MGISEQ-2000与NovaSeq6000的数据质量相近
我们制备了一系列cfDNA样品作为测试样品,以减少来比较测序仪之间的数据差异。将胰腺导管腺癌(PDAC)基因组DNA(gDNA)按0%、0.1%、0.5%、1%、5%和10%的浓度稀释到NA12878中,制备成模拟cfDNA样品。为了尽量减少试剂的影响,我们分别使用Illumina和MGI官方文库制备试剂盒制备”iLib”和“mLib”两套文库。然后,在NovaSeq6000上对“iLib”文库进行测序,在NovaSeq6000和MGISEQ-2000上对“mLib”文库进行测序,得到三个数据集:iLib- novaseq、mLib- novaseq和mLib- mgiseq。mLib-MGISEQ的测序错误率与iLib-NovaSeq相当,但明显低于mLib-NovaSeq,说明使用同一厂家生产的文库制备试剂盒和测序仪后制备文库和测序效果更好(图5)。
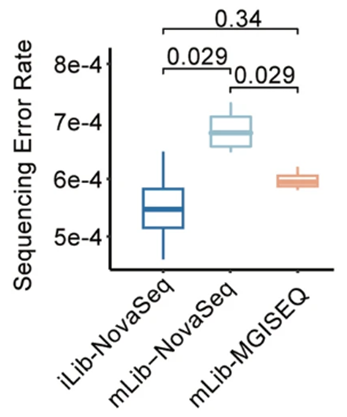
图5. 箱线图显示iLib-NovaSeq、mLib-NovaSeq和mLib-MGISEQ数据的测序错误率
3、MGISEQ‑2000与NovaSeq6000的甲基化测序水平高度吻合
mLib-MGISEQ和iLib-NovaSeq测序数据集的AMFs(平均甲基化分数)具有非常高的一致性,皮尔逊相关系数(PCC)为0.995,仅略低于iLib-NovaSeq和mLib-NovaSeq数据集的AMFs (PCC = 0.998)。(图6)
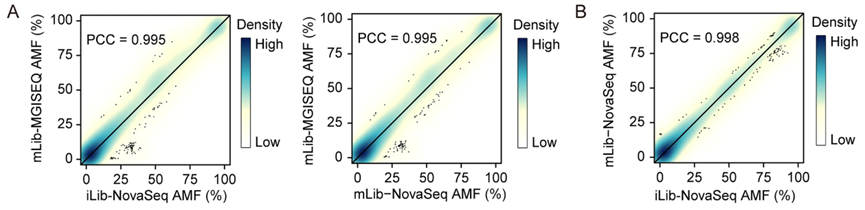
图6. iLib-NovaSeq与mLib-MGISEQ (A左)、mLib-NovaSeq与mLib-MGISEQ (A右)、iLib-NovaSeq与mLib-NovaSeq (B)之间的AMFs高度一致。
此外,MGISEQ-2000测的CpG甲基化水平与NovaSeq6000测的CpG甲基化水平一致,PCC为0.994(图7)。然而,在GC含量高的CpG位点,MGISEQ-2000的甲基化水平高于NovaSeq6000(图8)。
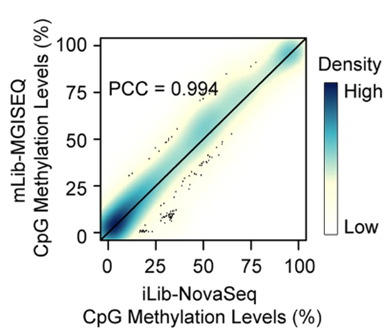
图7. mLib-MGISEQ与iLIb-NovaSeq测的甲基化水平高度一致
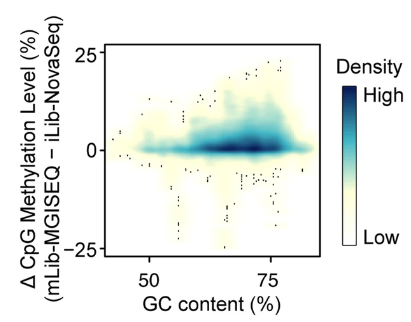
图8. mLib-MGISEQ和iLib-NovaSeq数据包之间CpG甲基化水平随GC含量的变化
4、与NovaSeq6000相比,MGISEQ‑2000在0.1%的肿瘤比例下显示出相当的癌症信号检测能力
检测平台的灵敏度对其临床应用很重要。结果显示,MGISEQ-2000在肿瘤比例为0.1%的情况下也能像NovaSeq6000一样显著地检测出癌症信号。(图9)
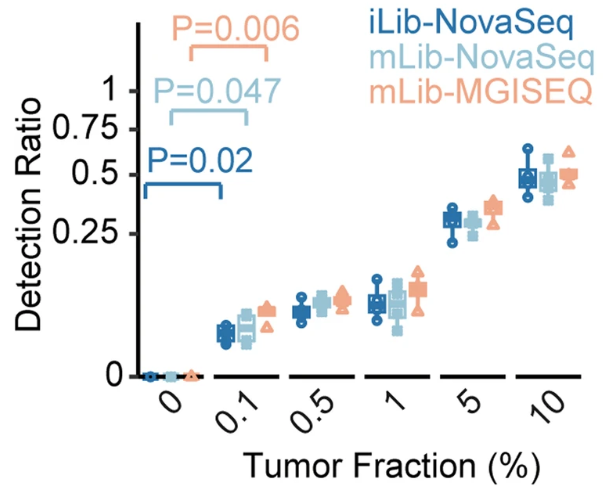
图9. iLib-NovaSeq、mLib-NovaSeq、mLib-MGISEQ的检出限(LOD)
5、在临床应用中,MGISEQ‑2000与NovaSeq6000表现相当
为了评估MGISEQ-2000在临床样本上的表现,我们检测了PDAC患者和健康人群的cfDNA构建的甲基化文库。在24个cfDNA样本中,只有两个(cfDNA16和cfDNA23)在mLib-MGISEQ和iLib-NovaSeq之间具有显著不同的预测分数,而其余的分数非常接近。此外,我们能够使用两个高度相似的阈值(iLib-NovaSeq数据为0.8952,mLib-MGISEQ数据为0.8956)以100%的准确率区分PDAC患者和健康人群的cfDNA样本。这些结果表明,MGISEQ-2000在临床检测中也具有高度的准确性(图10)。
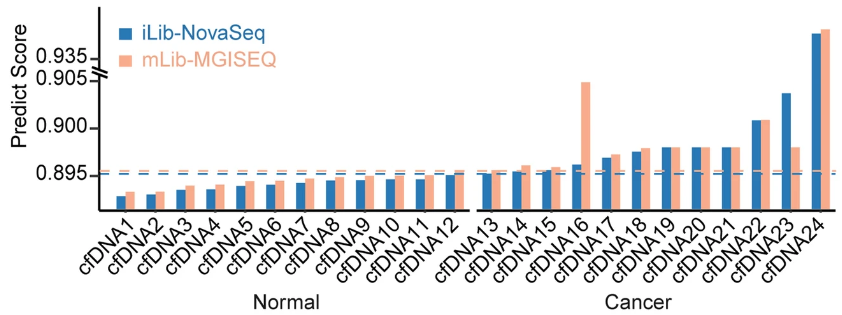
图10. 跨平台甲基化测序对PDCA的预测分数
研究结论:
我们采用MGISEQ-2000平台进行了亚硫酸氢盐靶向测序,以评估其潜在的应用前景。通过与广泛用于甲基化测序的NovaSeq6000平台进行头对头比较,对MGISEQ-2000的性能进行系统性测试。结果表明,MGISEQ-2000展现出与NovaSeq6000相似的测序质量、一致的甲基化水平、相当的癌症信号检测能力和准确的临床诊断结果,说明MGISEQ-2000可应用与临床检测DNA甲基化变化,特别是cfDNA甲基化检测。
参考资料:
[1] Sun J, Su M, Ma J, Xu M, Ma C, Li W, Liu R, He Q, Su Z. Cross-platform comparisons for targeted bisulfite sequencing of MGISEQ-2000 and NovaSeq6000. Clinical Epigenetics. 2023 Aug 14.









